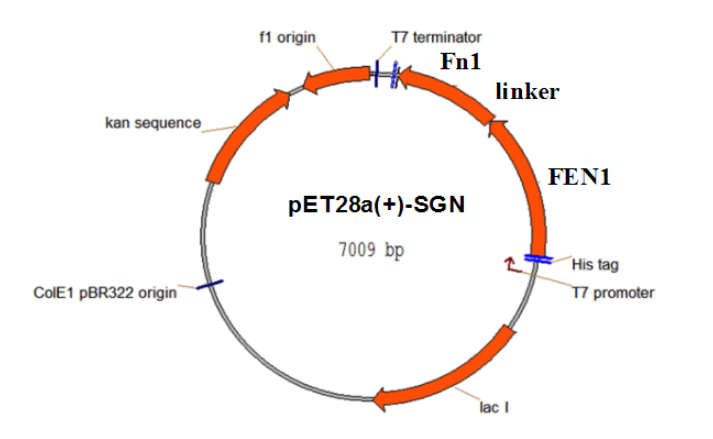
pET28a-SGN
pET28a-SGN
编号 | 载体名称 |
北京华越洋VECT75411 | pET28a-SGN |
pET28a-SGN载体基本信息:
启动子: | T7 |
复制子: | ColE1 ori |
终止子: | T7-ter |
质粒分类: | 大肠杆菌SGN基因编辑质粒 |
质粒大小: | 7009bp |
原核抗性: | 卡那霉素Kan |
pET28a-SGN载体图谱:
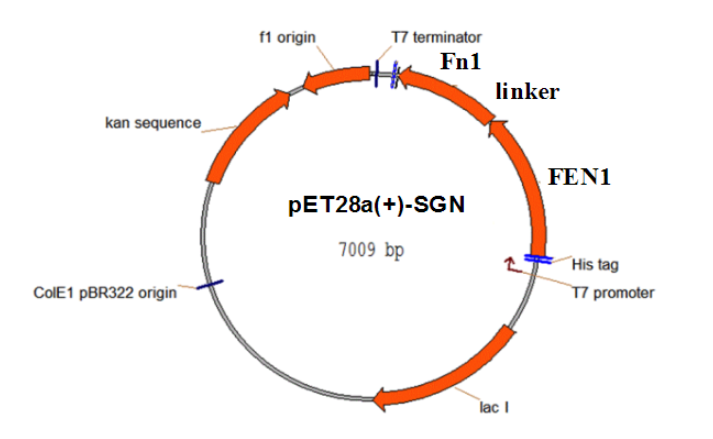
pET28a-SGN载体简介:
近日,南京大学医学院附属金陵医院的周国华教授、南京大学模式动物所的赵庆顺教授和朱敏生教授及他们的团队在GenomeBiology上报道了基因编辑的又一个全新的方法,结构引导的核酸内切酶技术SGN(structure-guided endonuclease)。这一新方法的突破在于,不同于以往的ZFN,TALEN, CRISPR-Cas, 甚至NgAgo等技术,SGN不受靶标序列限制。SGN中包含识别3’flap结构的内切酶FEN-1(flapendonuclease-1)和能够切割DNA链的Fok I (Fn1) 的切割结构域,人工合成的引导DNA(guide DNA)能够和靶标序列形成3’flap结构,该结构被SGN识别并结合后,SGN就可以对任何想要进行编辑的靶标DNA进行剪辑。
推测SGN产生大片段DNA删除的机制(Xu et al.,2016 [6])在GenomeBiology同期配发的ResearchHighlight[7]中,NIH的Shawn MBurgess教授总结了SGN的三个关键特征,
1.FEN-1与FokI切割结构域形成的融合蛋白(SGN)可以通过利用DNA寡聚体(向导DNA)去靶向突变特定基因位点。
2. 这种靶向突变方式倾向于产生大片段DNA的删除,删除片断可达几百至几千个碱基对。
3.SGN在斑马鱼胚胎中已显示出效果,表明这种基因编辑技术在模式动物中具备可行性。Burgess教授还宣称[7],在基因编辑上,SGN是一个令人激动的新选择,因为SGN非常灵活、简便,并且具有删除大片段DNA的潜力。这对于想要通过灭活某个基因以达到基因治疗或遗传改良效果的研究者来说,无疑是一大喜讯。此外,大片段DNA删除还可以防止在研究中出现的假阴性。
相较于评论专家及领域内研究者们的兴奋,该文章的作者们却表现得相当平静,赵庆顺教授对中国生物物理学会的采访者表示,这一项新技术才刚刚起步,SGN在很多方面都需要在后续工作中不断去优化和探索。
pET28a-SGN载体其他相关的载体:
| pTrc-Taq |
| pGEX-HRV3C |
| pET28a-ULP/SUMO |
| pET28a-TEV |
| pET28a-T4 lysozyme |
| pET28a-mMLV |
| pET22b-KOD |
| pBV220-EK |
| DL2000 |